Index >
Results for CNB 265692_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 -CCCATCAAGTACCTGCCTAATTTCTGTATGGCACACGCCAGAAATCAATAGAAGTTAAC
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 -CCCATCAAGTACCTGCCTAATTTCTGTATGGCACACGCCAGAAATCAATAGAAGTTAAC
265692_ENSG00000112238_HUMAN_9283_9748/1-473 -CCCATCAAGTACCTGCCTAATTTCTGTATGGCACACGCCAGAAATCAATAGTAGTTAAC
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 GCCCTTTCAGTACCTGCCTGATCCCAATCCGGCACACGCCGAGAGTCAATAGAAGTTAAC
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 AAGTCCTCCAAACAGTCCCCGTCCCTTTGTTTAATCTCCTTTACAATAAAGCCAAGGGGA
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 AAGTCCTCCAAACAGTCCCCGTCCCTTTGTTTAATCTCCTTTACAATAAAGCCAAGGGGA
265692_ENSG00000112238_HUMAN_9283_9748/1-473 AAGTCCTCCAAACAGTCTCCATCTCTTTGTTTAATCTCCTTTACAATAAAGCCAAGGGGA
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 AAGTCCTCTAAACTTTCCTGCCTCCTTTGTTGCA-CTAAGG-ACACAAAAGG--AGGAGG
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 GAGAATTAAGAATCTTTGAAGTAGACCAAGGCTCAAAGGAATCAG-CCAAGTAGACTTAA
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 GAGAATTAAGAATCTTTGAAGTAGACCAAGGCTCAAAGGAATCAG-CCAAGTAGACTTAA
265692_ENSG00000112238_HUMAN_9283_9748/1-473 GAGAATTAAAAATCTTTGAAGTAGATCAAGGCTCAAAGGAATCAG-CCAAGTAGACTTAA
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 GAAAATAAA-AAGCTTTGAAGTGCATTAAAGC-CAAAGAATGCTGGCCAAGTAGATTTAA
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 AGTAGTCACTTATTTCCCAAAACTGGTTTGTCGGTGAACAATAAAAGGAAGTAAA-ATTT
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 AGTAGTCACTTATTTCCCAAAACTGGTTTGTCGGTGAACAATAAAAGGAAGTAAA-ATTT
265692_ENSG00000112238_HUMAN_9283_9748/1-473 AGTAGTCACTTATTTCCCAAAACTGGTTTGTCGGGGAACAATAAAAGGAAGTAAA-ATTT
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 AGTAGCCACCGATTTCCAAAACCTTGCTTGAAAGGGAGGAATTGGAGAGGGCGAGGATTT
265692_ENSRNOG00000007683_RAT_9227_9692/1-473 ATGGAGAAATTATACAGTGGATTTGTCACTTAAAATATCGTAACTGTCTCAGGGACAATA
265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473 ATGGAGAAATTATACAGTGGATTTGTCACTTAAAATATCGTAACTGTCTCAGGGACAATA
265692_ENSG00000112238_HUMAN_9283_9748/1-473 ATGGAGAAATTATACAGTGGATTTGTCACTTAAAATATCGTAACTGTCTGGAGGACAATA
265692_SINFRUG00000152747_FUGU_5223_5688/1-473 ATGGATAAATTATACAGTGGATTTGTCAGTTAAAATATCGGAACTGTCTGGAGGACAAAG
265692_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 84.76
Mean single sequence MFE: -73.80
Consensus MFE: -48.96
Energy contribution: -52.03
Covariance contribution: 3.06
Mean z-score: -1.67
Structure conservation index: 0.66
SVM decision value: 0.13
SVM RNA-class probability: 0.596321
Prediction: RNA
######################################################################
>265692_ENSRNOG00000007683_RAT_9227_9692/1-473
CCCAUCAAGUACCUGCCUAAUUUCUGUAUGGCACACGCCAGAAAUCAAUAGAAGUUAACAAGUCCUCCAAACAGUCCCCGUCCCUUUGUUUAAUCUCCUUUACAAUAAAGCCAAGGGGAGAGAAUUAAGAAUCUUUGAAGUAGACCAAGGCUCAAAGGAAUCAGCCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGUGAACAAUAAAAGGAAGUAAAAUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUCAGGGACAAUA
........((.((((..((((((((((.((((....))))(....).))))))))))(((((((((((...((((((((....((((((((((......))).)))))))....)))))(.(((.((((..(((((((.((........))))))))).............((((......)))).)))).))).)....)))((((......)))).......))).(((.(((((......))))).)))...)))))))).......................)))).)).... ( -66.90)
>265692_ENSMUSG00000040478_MOUSE_2454_2920/1-473
CCCAUCAAGUACCUGCCUAAUUUCUGUAUGGCACACGCCAGAAAUCAAUAGAAGUUAACAAGUCCUCCAAACAGUCCCCGUCCCUUUGUUUAAUCUCCUUUACAAUAAAGCCAAGGGGAGAGAAUUAAGAAUCUUUGAAGUAGACCAAGGCUCAAAGGAAUCAGCCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGUGAACAAUAAAAGGAAGUAAAAUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUCAGGGACAAUA
........((.((((..((((((((((.((((....))))(....).))))))))))(((((((((((...((((((((....((((((((((......))).)))))))....)))))(.(((.((((..(((((((.((........))))))))).............((((......)))).)))).))).)....)))((((......)))).......))).(((.(((((......))))).)))...)))))))).......................)))).)).... ( -66.90)
>265692_ENSG00000112238_HUMAN_9283_9748/1-473
CCCAUCAAGUACCUGCCUAAUUUCUGUAUGGCACACGCCAGAAAUCAAUAGUAGUUAACAAGUCCUCCAAACAGUCUCCAUCUCUUUGUUUAAUCUCCUUUACAAUAAAGCCAAGGGGAGAGAAUUAAAAAUCUUUGAAGUAGAUCAAGGCUCAAAGGAAUCAGCCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGGGAACAAUAAAAGGAAGUAAAAUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUGGAGGACAAUA
........((..((((...((((((....(((....))))))))).....))))...))..((((((((.(((((((((((..((((.(((.((.((((..((((....((((..((((((..........(((((((.((........)))))))))........((((.((((......)))))))).)))))).....))))))))..))))...)).))).))))........)))))))........(((((.....))))).............)))).)))))))).... ( -73.80)
>265692_SINFRUG00000152747_FUGU_5223_5688/1-473
GCCCUUUCAGUACCUGCCUGAUCCCAAUCCGGCACACGCCGAGAGUCAAUAGAAGUUAACAAGUCCUCUAAACUUUCCUGCCUCCUUUGUUGCACUAAGGACACAAAAGGAGGAGGGAAAAUAAAAAGCUUUGAAGUGCAUUAAAGCCAAAGAAUGCUGGCCAAGUAGAUUUAAAGUAGCCACCGAUUUCCAAAACCUUGCUUGAAAGGGAGGAAUUGGAGAGGGCGAGGAUUUAUGGAUAAAUUAUACAGUGGAUUUGUCAGUUAAAAUAUCGGAACUGUCUGGAGGACAAAG
...(((((((.((....(((((.....((((((....)))).))...........(((((((((((((.....((((((.((((((((..(((......).))..)))))))).)))))).......((((((((.(((......((((........))))...)))..)))))))).(((.....(((((((..((((.((....)).))))..))))))).)))))))))))...((((((((........)))))))).)))))...)))))....)))))))))...... ( -87.60)
>consensus
_CCCAUCAAGUACCUGCCUAAUUUCUGUAUGGCACACGCCAGAAAUCAAUAGAAGUUAACAAGUCCUCCAAACAGUCCCCGUCCCUUUGUUUAAUCUCCUUUACAAUAAAGCCAAGGGGAGAGAAUUAAGAAUCUUUGAAGUAGACCAAGGCUCAAAGGAAUCAG_CCAAGUAGACUUAAAGUAGUCACUUAUUUCCCAAAACUGGUUUGUCGGGGAACAAUAAAAGGAAGUAAA_AUUUAUGGAGAAAUUAUACAGUGGAUUUGUCACUUAAAAUAUCGUAACUGUCUCAAGGACAAUA
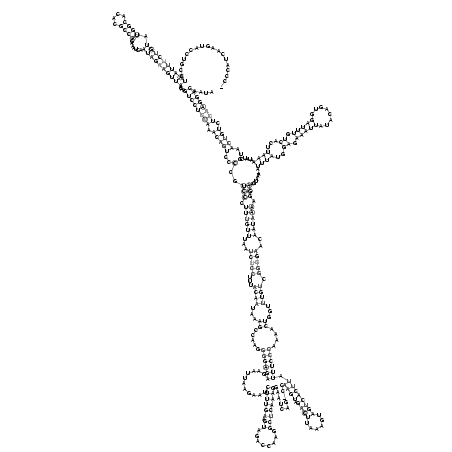
..................((((((((((.((((....)))).......))))))))))....((((((((.(((((..(..(((.((((((...(((((...((((...((.....((((((...........((((((.((........))))))))..........((((.((((......)))))))).))))))....))...)))).)))))..)))))).)))........((((..((.(((((........))))).))...)))).....)..))))).)))))))).... (-48.96 = -52.03 + 3.06)
265692_0.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004