Index >
Results for CNB 204490_3
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700 ATAAAATCAAAAGCACCTGCATCTTAAATGAAATTTCAATTGAAAAACTTAACAGCCCTT
204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700 ATATAATAAAAAGCACCTGCATTTTAAATGAAATTTCAATTGAAAAACTTAACAGCCCTT
204490_ENSRNOG00000022194_RAT_7638_8269/1-700 ATATAATAAAAAGCACCTGCATTTTAAATGAAATTTCAATTGAAAAACTTAACAGCCCTT
204490_ENSG00000152120_HUMAN_8890_9516/1-700 ATATAATAAAAAGCACCTGCATTTTAAATGAAATTTCAATTGAAAAACTTAACAGCCCTT
204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700 CAAATTGCGGAATTTAACGCAGACCAGTAAAGCTTAAATAACACTTTGCCTCTGCAGACC
204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700 CAAATTGCAGAATTTAACGCAGCCCAGTAAAGCTTAAATAACACTTTGCCAGCTCGGGCT
204490_ENSRNOG00000022194_RAT_7638_8269/1-700 CAAATTGCAGAATTTAACGCAGCCCAGTAAAGCTTAAATAACACTTTGCCTGCGCGGGCC
204490_ENSG00000152120_HUMAN_8890_9516/1-700 CAAATTGCAGAATTTAACGCAGCCCAGTAAAGCTTAAATAACACTTTGCCCCCGCAGGCT
204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700 GAAAGAGCGGANNNNNNNNNNNNNNNNNNNNNNNAGCTGGCCGGCGGTGAAGTAAGGAGG
204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700 GA--------------------------------------------------------GG
204490_ENSRNOG00000022194_RAT_7638_8269/1-700 GA--------------------------------------------------------GG
204490_ENSG00000152120_HUMAN_8890_9516/1-700 GA--------------------------------------------------------GG
204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700 GGTGGTTGGCGTTCAAGGTGACTTGATTGTCTCATGTGGTAAAATTATTTACAATTAAGG
204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700 GGA--TTCGCATTCAAGGTGACTTGATTGTGTCGCATGGTGAAATTATTTACAATTAAGG
204490_ENSRNOG00000022194_RAT_7638_8269/1-700 GGA--TTCGCATTCAAGGTGACTTGATTGTGTCGCATGGTGAAATTATTTACAATTAAGG
204490_ENSG00000152120_HUMAN_8890_9516/1-700 GGT--TT-GCATTCAAGGTGACTTGATTGTGTCGCATGGTGAAATTATTTACAATTAAGG
204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700 AATTTCTCCTGAGAGCCACATTGGATTTGCCGCCG---AGCACCTATGCGGGCGTTTGGG
204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700 AATTTCTCTGGAGGGCTGCAGAGAGCTTGCTGCCCCCCGCCACCTCTGCAGGCATATGGT
204490_ENSRNOG00000022194_RAT_7638_8269/1-700 AATTTCTCTGGAGGGCTGCAGAGAGCTTGCTGNNNNNNNNN---TCTGCAGGCATATGGT
204490_ENSG00000152120_HUMAN_8890_9516/1-700 AATTTCTCTGGAGGGCTGCAGAATATTTGCTGTTCCACAACACCTATGCAGGCATATGGT
204490_3.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 78.35
Mean single sequence MFE: -71.88
Consensus MFE: -44.34
Energy contribution: -49.40
Covariance contribution: 5.06
Mean z-score: -2.38
Structure conservation index: 0.62
SVM decision value: 1.67
SVM RNA-class probability: 0.971070
Prediction: RNA
######################################################################
>204490_ENSDARG00000005719_ZEBRAFISH_1509_2198/1-700
AUAAAAUCAAAAGCACCUGCAUCUUAAAUGAAAUUUCAAUUGAAAAACUUAACAGCCCUUCAAAUUGCGGAAUUUAACGCAGACCAGUAAAGCUUAAAUAACACUUUGCCUCUGCAGACCGAAAGAGCGGANNNNNNNNNNNNNNNNNNNNNNNAGCUGGCCGGCGGUGAAGUAAGGAGGGGUGGUUGGCGUUCAAGGUGACUUGAUUGUCUCAUGUGGUAAAAUUAUUUACAAUUAAGGAAUUUCUCCUGAGAGCCACAUUGGAUUUGCCGCCGAGCACCUAUGCGGGCGUUUGGG
..........((((.(((((((.((((.(((....))).)))).............(((((...(((((........)))))....................((((..(((((((....(....).)))))........................(((....)))))..))))..)))))((((.((((((.(((((....)))))..((((.(((((((..((((......)))).((((.....))))....))))))).))))....)))))).)))).))))))).))))... ( -85.40)
>204490_ENSMUSG00000039186_MOUSE_8899_9531/1-700
AUAUAAUAAAAAGCACCUGCAUUUUAAAUGAAAUUUCAAUUGAAAAACUUAACAGCCCUUCAAAUUGCAGAAUUUAACGCAGCCCAGUAAAGCUUAAAUAACACUUUGCCAGCUCGGGCUGAGGGGAUUCGCAUUCAAGGUGACUUGAUUGUGUCGCAUGGUGAAAUUAUUUACAAUUAAGGAAUUUCUCUGGAGGGCUGCAGAGAGCUUGCUGCCCCCCGCCACCUCUGCAGGCAUAUGGU
...........((((...((.((((((.(((....))).))))))..(((..(((((((((....(((.((((((....(((((((((...((..............))..))).))))))...))))))(((.(((((....))))).)))...))).((.((((((.((((....)))).)))))).)))))))))))..))).)).))))(((....(((.........)))....))) ( -72.14)
>204490_ENSRNOG00000022194_RAT_7638_8269/1-700
AUAUAAUAAAAAGCACCUGCAUUUUAAAUGAAAUUUCAAUUGAAAAACUUAACAGCCCUUCAAAUUGCAGAAUUUAACGCAGCCCAGUAAAGCUUAAAUAACACUUUGCCUGCGCGGGCCGAGGGGAUUCGCAUUCAAGGUGACUUGAUUGUGUCGCAUGGUGAAAUUAUUUACAAUUAAGGAAUUUCUCUGGAGGGCUGCAGAGAGCUUGCUGNNNNNNNNNUCUGCAGGCAUAUGGU
............((....)).((((((.(((....))).))))))..(((..(((((((((....(((.((((((....(.((((.(((..((..............)).)))..)))).)...))))))(((.(((((....))))).)))...))).((.((((((.((((....)))).)))))).)))))))))))..))).((((((..............))))))....... ( -66.48)
>204490_ENSG00000152120_HUMAN_8890_9516/1-700
AUAUAAUAAAAAGCACCUGCAUUUUAAAUGAAAUUUCAAUUGAAAAACUUAACAGCCCUUCAAAUUGCAGAAUUUAACGCAGCCCAGUAAAGCUUAAAUAACACUUUGCCCCCGCAGGCUGAGGGGUUUGCAUUCAAGGUGACUUGAUUGUGUCGCAUGGUGAAAUUAUUUACAAUUAAGGAAUUUCUCUGGAGGGCUGCAGAAUAUUUGCUGUUCCACAACACCUAUGCAGGCAUAUGGU
...............((((((((((((.(((....))).)))))).....((((((..((((((((....)))))...(((((((.........(((((((((((.(((((((.(.....).))))...(((.(((((....))))).)))...))).))))...))))))).......(((.....)))...))))))).))).....))))))............))))))........ ( -63.50)
>consensus
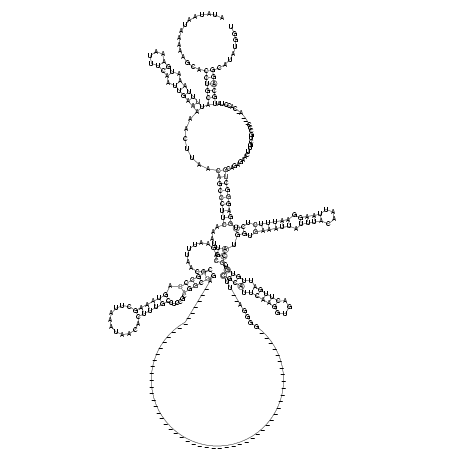
AUAUAAUAAAAAGCACCUGCAUUUUAAAUGAAAUUUCAAUUGAAAAACUUAACAGCCCUUCAAAUUGCAGAAUUUAACGCAGCCCAGUAAAGCUUAAAUAACACUUUGCCUCCGCAGGCCGA________________________________________________________GGGGA__UUCGCAUUCAAGGUGACUUGAUUGUGUCGCAUGGUGAAAUUAUUUACAAUUAAGGAAUUUCUCUGGAGGGCUGCAGAGAACUUGCUGCCC___A_CACCUAUGCAGGCAUAUGGU
...............((((((((((((.(((....))).)))))).......(((((((((....(((...........((((((.((((((...........))))))......))))))..................................................................((((.(((((....))))).))))..))).((.((((((.((((....)))).)))))).)))))))))))............................))))))........ (-44.34 = -49.40 + 5.06)
204490_3.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004