Index >
Results for CNB 134297_12-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 A--GCAGGTGTCTACAGGCGTCTCCCCA--TCTAGATAA-AACTAATAAAATCGCCCATC
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 A--GCAGGTGTCTGCAGCCATCTCCCCA--TCCAGATAA-AACTAATAAAATCGCCCATC
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 AATAAAGATGTTCACGGGATACGACCCAGCCCCAAATAAGACCTTATCAGGAC-AGCATC
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 CATAGAAAGTTTAAGATGAAATGGATTG--GGCTTTTTATAAATACTCATCTC-ACCCTG
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 A--GCAGGTGTCTACAGGCGTCTCCCCA--TCTAGATAA-AACTAATAAAATCGCCCATC
* * * * * * * * * * *
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 CAAC-ACTGATGTCAATATTACT-TTAAATTA-CACGAAATCAAATTTA--TTTTTAATT
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 CAAC-ACTGATGTCAATATTACT-TTAAATTA-CACAAAATCAAATTTA--TTTTTAATT
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 CAAC-TCTGATG-CTTCCTCAGC-AGCACAGA-ATCAAATTC-ATTTTT--TAATTAGGC
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 AAGCTTGCTTTTTAAACGTTAATCATAAATAACCATTAGTTCCAATAAAACATTTTAATT
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 CAAC-ACTGATGTCAATATTACT-TTAAATTA-CACGAAATCAAATTTA--TTTTTAATT
* * * * * * * * ** * * ***
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 AGC-AAATTAATAGGCGGCAGTTGAGGGTTTTAATTACTCAATTAACTTCACGCAAGTTA
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 AGC-AAATTAATAGGCGGCAGTTGAGGGTTTTAATTACTCAATTAACTTCACGCAAGTTA
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 GGCTAAATTAATAGGCGGCAGTTTAGGAGTTTAATTACTCAATTAACTTCACGCACGTTA
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 AAA-AAATCAATATACTACTTTTGAAGGTCTTAATTGCTGAATTAACGTAACGTGAATTC
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 AGC-AAATTAATAGGCGGCAGTTGAGGGTTTTAATTACTCAATTAACTTCACGCAAGTTA
**** **** * * ** * * ****** ** ******* * *** **
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 ATTTTTAACTATCTAAATTAACTTGCACATTACTCGATTTGCTGATAATTACAGAATTAA
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 ATTTTTAACTATCTAAATTAACTTGCGCATTACTCGATTTGCTGATAATTACAGAATTAA
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 ATTTTTAACTATCTAAATTAGGTTGCACATTATTCGATTTGATGATAATTATAGAATTAA
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 ATTTGTAGCTATCATAACTGAGCTGCACATTAGTCGATGTACAGATAATTAGAGTGTGAA
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 ATTTTTAACTATCTAAATTAACTTGCACATTACTCGATTTGCTGATAATTACAGAATTAA
**** ** ***** ** * *** ***** ***** * ******** ** * **
134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004 ATCTAAATTAATTGTTGGAGGTGATTTGATCCGCTGCTGAACTGGATGCAATTCCAATTA
134297_ENSG00000164853_HUMAN_5679_7549/1-2004 ATCTAAATTAATTGTTGGAGGTGATTTGATCCGCTGCTGAACTGGATGAGATTCCAATTA
134297_SINFRUG00000149320_FUGU_3658_5564/1-2004 ATCAAAATTAATTGTTTTGGATGATTTG-------GGTGAACTAGATGCAGTTCCAATTA
134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004 ACGTAAATTAATTGTTGAAAGTCATT-----------TGATCCACAAACGGAACCAATTA
134297_ENSRNOG00000001284_RAT_5363_7190/1-2004 ATCTAAATTAATTGTTGGAGGTGATTTGATCCGCTGCTGAACTGGATGCAATTCCAATTA
* ************ * *** *** * * *******
134297_12-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 73.22
Mean single sequence MFE: -63.85
Consensus MFE: -24.84
Energy contribution: -27.48
Covariance contribution: 2.64
Mean z-score: -2.16
Structure conservation index: 0.39
SVM decision value: 1.54
SVM RNA-class probability: 0.963496
Prediction: RNA
######################################################################
>134297_ENSMUSG00000029546_MOUSE_5334_7205/1-2004
AGCAGGUGUCUACAGGCGUCUCCCCAUCUAGAUAAAACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACGAAAUCAAAUUUAUUUUUAAUUAGCAAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGCAAUUCCAAUUA
(((((.((((((..((.(....)))...)))))).........((((((((..(((((((.((((.((....................)).))))((((((..(((((((....(((((.....(((((((((((((..((((((....))))))..))..((((((((((((..........))))))))))))......))))).)))))).)))))....)))))))..))))))...)))))))))))))))......)))))....((((......)))).... ( -68.25)
>134297_ENSG00000164853_HUMAN_5679_7549/1-2004
AGCAGGUGUCUGCAGCCAUCUCCCCAUCCAGAUAAAACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACAAAAUCAAAUUUAUUUUUAAUUAGCAAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCGCAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGAGAUUCCAAUUA
.((((....)))).(..(((((...((((((............((((((((..(((((((.((((..........................))))((((((..(((((((....(((((.....(((((((((((((..((((((....))))))..)).(((((((((((((..........))))))))))))).....))))).)))))).)))))....)))))))..))))))...)))))))))))))))..............)))))))))))..)..... ( -69.64)
>134297_SINFRUG00000149320_FUGU_3658_5564/1-2004
AAUAAAGAUGUUCACGGGAUACGACCCAGCCCCAAAUAAGACCUUAUCAGGACAGCAUCCAACUCUGAUGCUUCCUCAGCAGCACAGAAUCAAAUUCAUUUUUUAAUUAGGCGGCUAAAUUAAUAGGCGGCAGUUUAGGAGUUUAAUUACUCAAUUAACUUCACGCACGUUAAUUUUUAACUAUCUAAAUUAGGUUGCACAUUAUUCGAUUUGAUGAUAAUUAUAGAAUUAAAUCAAAAUUAAUUGUUUUGGAUGAUUUGGGUGAACUAGAUGCAGUUCCAAUUA
.........(((((((((......)))....((((((....(((....)))....(((((((.((((.((((........))))))))...............((((((((((((.((((((((..((((.((((...((((......))))....)))).).)))..))))))))....(((.......)))))))).........((((((((..((....))..))))))))....)))))))..))))))))))))))))))).................. ( -67.00)
>134297_ENSDARG00000018024_ZEBRAFISH_6522_8353/1-2004
CAUAGAAAGUUUAAGAUGAAAUGGAUUGGGCUUUUUAUAAAUACUCAUCUCACCCUGAAGCUUGCUUUUUAAACGUUAAUCAUAAAUAACCAUUAGUUCCAAUAAAACAUUUUAAUUAAAAAAUCAAUAUACUACUUUUGAAGGUCUUAAUUGCUGAAUUAACGUAACGUGAAUUCAUUUGUAGCUAUCAUAACUGAGCUGCACAUUAGUCGAUGUACAGAUAAUUAGAGUGUGAAACGUAAAUUAAUUGUUGAAAGUCAUUUGAUCCACAAACGGAACCAAUUA
.(((((((((((((..(.....)..)))))))))))))..................((((....))))......((((........)))).....(((((......((((((((((((.....((..(((((.(((..((..............((((((.(((...))).))))))..(((((((..........)))))))))..))).).))))..)))))))))))))).....((..(((((.((.(....).)).)))))..))....)))))...... ( -46.10)
>134297_ENSRNOG00000001284_RAT_5363_7190/1-2004
AGCAGGUGUCUACAGGCGUCUCCCCAUCUAGAUAAAACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACGAAAUCAAAUUUAUUUUUAAUUAGCAAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGCAAUUCCAAUUA
(((((.((((((..((.(....)))...)))))).........((((((((..(((((((.((((.((....................)).))))((((((..(((((((....(((((.....(((((((((((((..((((((....))))))..))..((((((((((((..........))))))))))))......))))).)))))).)))))....)))))))..))))))...)))))))))))))))......)))))....((((......)))).... ( -68.25)
>consensus
A__GCAGGUGUCUACAGGCAUCUCCCCA__UCCAGAUAA_AACUAAUAAAAUCGCCCAUCCAAC_ACUGAUGUCAAUAUUACU_UUAAAUUA_CACAAAAUCAAAUUUA__UUUUUAAUUAGC_AAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUUAAAUCUAAAUUAAUUGUUGGAGGUGAUUUGAUCCGCUGCUGAACUGGAUGCAAUUCCAAUUA
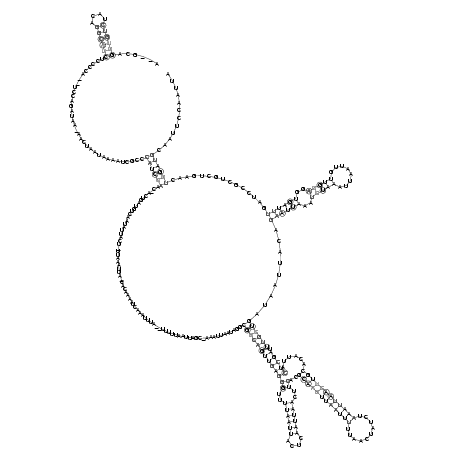
......((((((....))))))..................................((((((.........................................................................(((((((((((((..((((((....))))))..))..((((((((((((..........))))))))))))......))))).)))))).........((((((..(((((........)))))..))))))...............))))))............ (-24.84 = -27.48 + 2.64)
134297_12-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004