Index >
Results for CNB 134270_20-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687 GAAACCGTGCCTCTGGCGGAAAA-TGAGCATGTGCAATCAAGTAGACGTCACTGACCCCA
134270_ENSG00000164853_HUMAN_5374_7839/1-2687 GGGTCGGGGGCGGCCGGAGCGCC-TCC-CCC-TCTGGC---CCGCCAGCCCCGGGGCCGT
134270_SINFRUG00000149320_FUGU_3315_5852/1-2687 TTAAGAGGA--TTTTGGAGCGCCTTCCTCCG-GATAACTGAATTTCACAGGCGTCGCCAA
134270_ENSRNOG00000001284_RAT_5040_7483/1-2687 -GGACAAGG--TGTTGAGGCACC-CAC-ACT-TCTAAC---ACCACCAAACTC-GGTTGC
134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687 GGGACAAGG--TGTTGAGGCACC-CAC-ACT-TCCAAT---ACCACCGAACTCGGGTTGC
* *
134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687 TTAACAGCAGGTGTTCAGATGTAACG-CCTA--ACCAAA-CA-AACTAATCAAAGCGCCC
134270_ENSG00000164853_HUMAN_5374_7839/1-2687 CTAA--GCAGGTGTCTGCAGCCATCTCCCCA--TCCAGATAA-AACTAATAAAATCGCCC
134270_SINFRUG00000149320_FUGU_3315_5852/1-2687 CTAAATAAAGATGTTCACGGGATACGACCCAGCCCCAAATAAGACCTTATCAGGAC-AGC
134270_ENSRNOG00000001284_RAT_5040_7483/1-2687 CTAA--GCAGGTGTCTACAGGCGTCTCCCCA--TCTAGATAA-AACTAATAAAATCGCCC
134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687 CTAA--GCAGGTGTCTACAGGCGTCTCCCCA--TCTAGATAA-AACTAATAAAATCGCCC
*** ** *** * ** * * * * * * ** ** * * *
134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687 ATCCAACACCGACGCCCGCATCAC-TAACATTACATAAAATCAAATTCATTTTTAATTAG
134270_ENSG00000164853_HUMAN_5374_7839/1-2687 ATCCAACACTGATGTCAATATTACTTTAAATTACACAAAATCAAATTTATTTTTAATTA-
134270_SINFRUG00000149320_FUGU_3315_5852/1-2687 ATCCAACTCTGATG-CTTCCTCAGCAGCACAGAATCAAATTC-ATTTTTTAATTAGGCG-
134270_ENSRNOG00000001284_RAT_5040_7483/1-2687 ATCCAACACTGATGTCAATATTACTTTAAATTACACGAAATCAAATTTATTTTTAATTA-
134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687 ATCCAACACTGATGTCAATATTACTTTAAATTACACGAAATCAAATTTATTTTTAATTA-
******* * ** * * * * * ** ** * ** * ***
134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687 GCTAGCAAATTAATACGCAACAGCTGAGGAGTTTAATTACTCAATTAACTTCACGCGCGT
134270_ENSG00000164853_HUMAN_5374_7839/1-2687 GC----AAATTAATAGGCGGCAGTTGAGGGTTTTAATTACTCAATTAACTTCACGCAAGT
134270_SINFRUG00000149320_FUGU_3315_5852/1-2687 GCT---AAATTAATAGGCGGCAGTTTAGGAGTTTAATTACTCAATTAACTTCACGCACGT
134270_ENSRNOG00000001284_RAT_5040_7483/1-2687 GC----AAATTAATAGGCGGCAGTTGAGGGTTTTAATTACTCAATTAACTTCACGCAAGT
134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687 GC----AAATTAATAGGCGGCAGTTGAGGGTTTTAATTACTCAATTAACTTCACGCAAGT
** ********* ** *** * *** ************************* **
134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687 TAATTTTTAACTATCTAAATTAAGTTGCACATTATTCGATTTGATGATAATTATAGAATT
134270_ENSG00000164853_HUMAN_5374_7839/1-2687 TAATTTTTAACTATCTAAATTAACTTGCGCATTACTCGATTTGCTGATAATTACAGAATT
134270_SINFRUG00000149320_FUGU_3315_5852/1-2687 TAATTTTTAACTATCTAAATTAGGTTGCACATTATTCGATTTGATGATAATTATAGAATT
134270_ENSRNOG00000001284_RAT_5040_7483/1-2687 TAATTTTTAACTATCTAAATTAACTTGCACATTACTCGATTTGCTGATAATTACAGAATT
134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687 TAATTTTTAACTATCTAAATTAACTTGCACATTACTCGATTTGCTGATAATTACAGAATT
********************** **** ***** ******** ********* ******
134270_20-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 73.45
Mean single sequence MFE: -65.68
Consensus MFE: -24.84
Energy contribution: -27.72
Covariance contribution: 2.88
Mean z-score: -1.96
Structure conservation index: 0.38
SVM decision value: 0.87
SVM RNA-class probability: 0.870174
Prediction: RNA
######################################################################
>134270_ENSDARG00000020961_ZEBRAFISH_5965_8474/1-2687
GAAACCGUGCCUCUGGCGGAAAAUGAGCAUGUGCAAUCAAGUAGACGUCACUGACCCCAUUAACAGCAGGUGUUCAGAUGUAACGCCUAACCAAACAAACUAAUCAAAGCGCCCAUCCAACACCGACGCCCGCAUCACUAACAUUACAUAAAAUCAAAUUCAUUUUUAAUUAGGCUAGCAAAUUAAUACGCAACAGCUGAGGAGUUUAAUUACUCAAUUAACUUCACGCGCGUUAAUUUUUAACUAUCUAAAUUAAGUUGCACAUUAUUCGAUUUGAUGAUAAUUAUAGAAUU
....((((.......)))).........(((((((((...(..(.((((.(((..........)))..((((((..((((...(((......................)))..)))).)))))))))).)..)................................((((((.((.(((.(((((((..(((..(....).((((((.((((....))))))))))..)))..)))))))....))).)).))))))))))))))).((((....((((....))))..)))). ( -52.85)
>134270_ENSG00000164853_HUMAN_5374_7839/1-2687
GGGUCGGGGGCGGCCGGAGCGCCUCCCCCUCUGGCCCGCCAGCCCCGGGGCCGUCUAAGCAGGUGUCUGCAGCCAUCUCCCCAUCCAGAUAAAACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACAAAAUCAAAUUUAUUUUUAAUUAGCAAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCGCAUUACUCGAUUUGCUGAUAAUUACAGAAUU
(((.(((((((((((((((.(......))))))))).....)))))((((........((((....))))........))))......................)))))........(((...............((((((...........))))))......(((((((((((....((.(..(((((((...........)))))))..).)).(((((((((((((..........)))))))))))))........))))))))))).....))).... ( -78.49)
>134270_SINFRUG00000149320_FUGU_3315_5852/1-2687
UUAAGAGGAUUUUGGAGCGCCUUCCUCCGGAUAACUGAAUUUCACAGGCGUCGCCAACUAAAUAAAGAUGUUCACGGGAUACGACCCAGCCCCAAAUAAGACCUUAUCAGGACAGCAUCCAACUCUGAUGCUUCCUCAGCAGCACAGAAUCAAAUUCAUUUUUUAAUUAGGCGGCUAAAUUAAUAGGCGGCAGUUUAGGAGUUUAAUUACUCAAUUAACUUCACGCACGUUAAUUUUUAACUAUCUAAAUUAGGUUGCACAUUAUUCGAUUUGAUGAUAAUUAUAGAAUU
....(((((.(((((.((..........(((((.((..((((....(((...)))....))))..)).)))))..(((......))).)).))))).....(((....)))..((((((.......))))))))))).(((((...((((...)))).........((((..((..((((((((..((((.((((...((((......))))....)))).).)))..))))))))....))..)))).....))))).(((((.......))))).............. ( -65.30)
>134270_ENSRNOG00000001284_RAT_5040_7483/1-2687
GGACAAGGUGUUGAGGCACCCACACUUCUAACACCACCAAACUCGGUUGCCUAAGCAGGUGUCUACAGGCGUCUCCCCAUCUAGAUAAAACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACGAAAUCAAAUUUAUUUUUAAUUAGCAAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUU
.((((.((((((((((((((...............(((......)))(((....))))))))))...((((..........(((......))).......))))....)))))))..)))).......................................(((((((((((....((.(..(((((((...........)))))))..).))..((((((((((((..........)))))))))))).........)))))))))))............ ( -64.63)
>134270_ENSMUSG00000029546_MOUSE_5013_7498/1-2687
GGGACAAGGUGUUGAGGCACCCACACUUCCAAUACCACCGAACUCGGGUUGCCUAAGCAGGUGUCUACAGGCGUCUCCCCAUCUAGAUAAAACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACGAAAUCAAAUUUAUUUUUAAUUAGCAAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUU
..((((.((((((((((((((................(((....)))..(((....))))))))))...((((..........(((......))).......))))....)))))))..)))).......................................(((((((((((....((.(..(((((((...........)))))))..).))..((((((((((((..........)))))))))))).........)))))))))))............ ( -67.13)
>consensus
GGGACAGGG__UGUUGAGGCACC_UAC_CCU_UCUAAC___ACCACCGAACCCGGGCCGACUAA__GCAGGUGUCUACAGGCAUCUCCCCA__UCCAGAUAA_AACUAAUAAAAUCGCCCAUCCAACACUGAUGUCAAUAUUACUUUAAAUUACACAAAAUCAAAUUUAUUUUUAAUUA_GC____AAAUUAAUAGGCGGCAGUUGAGGGUUUUAAUUACUCAAUUAACUUCACGCAAGUUAAUUUUUAACUAUCUAAAUUAACUUGCACAUUACUCGAUUUGCUGAUAAUUACAGAAUU
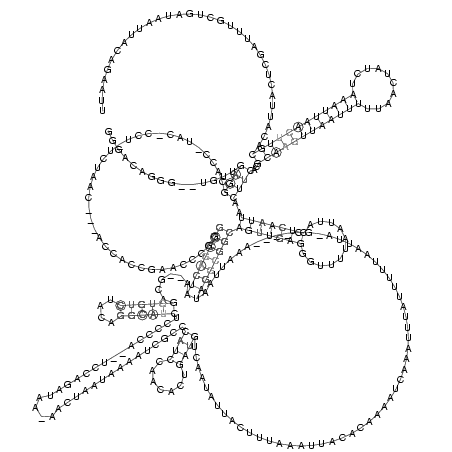
...............((((....................................((((((((......((((((....))))))..................................((((.......))))...........................................................))))))))(((((((...........)))))))..))))..((((((((((((..........))))))))))))................................ (-24.84 = -27.72 + 2.88)
134270_20-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004